Los evolucionistas presentan con
frecuencia la adquisición por parte de las bacterias de
resistencia a los antibióticos como una demostración de
cambio evolutivo. Sin embargo, el análisis molecular de los
mecanismos genéticos que llevan a la resistencia a los
antibióticos no da soporte a esta suposición tan
extendida. Muchas bacterias se convierten en resistentes por la
adquisición de genes procedentes de plásmidos o de
transposones mediante transferencia horizontal de genes. Sin embargo,
la transferencia horizontal no explica el origen de los genes de
resistencia, solo su difusión entre las bacterias. Las
mutaciones, por su parte, pueden explicar el surgimiento de la
resistencia a los antibióticos dentro del mundo bacteriano, pero
implican procesos mutacionales que son contrarios a las predicciones de
la evolución. Lo que hacen estas mutaciones es reducir o
eliminar la función de las proteínas de transporte o de
las porinas, las afinidades de enlace de las proteínas, las
actividades de los enzimas, la fuerza motriz protónica, o de los
sistemas de control y regulación. En tanto que dichas mutaciones
pueden considerarse como «beneficiosas» en cuanto que
aumentan la tasa de supervivencia de las bacterias en presencia del
antibiótico, implican procesos de mutación que no
proporcionan un mecanismo genético para una «descendencia
común con modificación». Asimismo, es frecuente que
con estas mutaciones esté asociado algún coste de
«capacidad relativa», aunque las mutaciones inversas pueden
en su momento recuperar la mayor parte del coste, si no todo, en el
caso de algunas bacterias. Pero se da un verdadero coste
biológico con la pérdida de sistemas o funciones
celulares preexistentes. Esta pérdida de actividad celular no
puede presentarse de forma legítima como un medio
genético de prueba de evolución.
Introducción
Debido a su
gran velocidad de reproducción,
facilidad de
análisis en laboratorio y la amplia diversidad que se puede
obtener de mutantes
generados en
laboratorio, las bacterias se han descrito como
un
excelente modelo para estudiar el proceso de la evolución
(Mortlock, 1984). La
adquisición de resistencia a un antibiótico
específico proporciona un evidente
beneficio a la bacteria cuando se expone a dicho antibiótico. De
modo que la
adquisición de resistencia a los antibióticos se cita
generalmente como ejemplo
de «cambio evolutivo», y ha llegado a ser un ejemplo
popular de lo que se
denomina «evolución en una cápsula de Petri».
Miller (1999) se refiere al
desarrollo de la resistencia a los antibióticos como un ejemplo
de la «fuerza
creadora» de la evolución. Barlow y Hall (2002) se
refieren
a ella como «la singular
oportunidad de observar procesos evolutivos a lo largo de unas pocas
décadas en
lugar de los varios milenios que son generalmente necesarios para estos
procesos» (p. 314).
Con frecuencia la evolución se describe meramente como
«cambio» o «cambio en frecuencia de los genes a lo
largo del tiempo» (Dillon,
1978; Johnson, 2000; Patterson, 1978), y los evolucionistas han
mantenido casi
universalmente que cualquier cambio en el genotipo (o incluso en el
fenotipo)
es un «cambio evolutivo». Como tal, cualquier cambio
biológico en un organismo,
incluyendo la resistencia a los antibióticos, concordaría
con esta definición.
Sin embargo, el mero cambio biológico también es
congruente con un modelo de
creación, y por ello esta definición «todo
terreno» no es fácilmente
distinguible del concepto creacionista. Esta definición tampoco
define la clase
de cambio (como deletéreo frente a beneficioso), y por ello no
proporciona
ningún valor predictivo a la teoría.
Además, cualquier cambio que parezca
proporcionar una adaptación
pretendidamente «beneficiosa» se considera generalmente
como una fuerza
impulsora de evolución. Desde luego, hay mutaciones, como la
resistencia a los
antibióticos, que pueden ser beneficiosas porque pueden dar al
organismo una mayor
capacidad para sobrevivir bajo condiciones medioambientales muy
específicas.
Así, por lo general, los evolucionistas concluyen que los
ejemplos genéticos de
«cambio evolutivo» son abundantes, y que los creacionistas
se ven obligados a
negar esta evidencia tan ampliamente observada.
Sin embargo, la teoría de la
evolución propone que
toda la
vida en la tierra tuvo un origen común. De ahí, toda la
vida comparte
un
ancestro evolutivo común del que ha descendido, esto es, la
«descendencia
común» de toda la vida. En una declaración a modo
de resumen, Darwin
(1936)
dice que «la teoría de la descendencia con
modificación abarca a todos
los
miembros de la misma clase principal o reino ... todos los animales y
plantas
descienden de algún prototipo» (p. 370). Por tanto,
mediante esta
«descendencia
con modificación» global y común, la teoría
de la evolución pretende
dar cuenta
del origen y de la diversidad de todo el desarrollo biológico en
la
tierra. Así, la «descendencia común con
modificación» proporciona una
definición más apropiada
y funcional de la teoría de la evolución, y este
artículo se refiere a
la
evolución en este contexto. Esta definición implica
también varias
«predicciones» respecto a las clases de cambio
genético necesario para
una
descendencia evolutiva común (predicciones que contrastan
acusadamente
con las
predicciones de un «modelo creacionista»). Estos cambios
han de
proporcionar
algo más que meros cambios en el fenotipo; han de proporcionar
un
mecanismo
genético que explique el origen de las funciones y actividades
celulares (esto
es, sistemas de regulación, sistemas de transporte,
especificidad
enzimática,
afinidad de enlaces de las proteínas, etc.).
Los cambios genéticos que reducen o
eliminan alguno de estos
sistemas celulares no proporcionan un mecanismo genético para la
«descendencia
común con modificación». Al contrario, estos
cambios son en realidad contrarios
a tal descendencia, al reducir o eliminar un sistema preexistente de
complejidad biológica (una inversión del «descenso
con modificación»). Por ello,
estos cambios genéticos no sirven como ejemplo de un mecanismo
genético para la
adquisición «evolutiva» del vuelo por parte de
organismos no voladores, de
conocimiento por parte de organismos no cognoscitivos, de
fotosíntesis por
parte de organismos no fotosintetizadores, etc. En cambio, la
teoría de la
evolución necesita que se hayan dado estos
acontecimientos, y necesita
unas mutaciones capaces de producir estos cambios genéticos. Por
ello, las
predicciones de la evolución precisan de ciertos tipos
específicos de cambios,
no meramente las mutaciones conocidas como «beneficiosas».
Así, a pesar de
las grandes pretensiones que hacen, es imprescindible plantear si la
adquisición de la resistencia a los antibióticos es un
ejemplo válido de cambio
evolutivo que dé sustento a las predicciones de la teoría
evolucionista (esto
es, la teoría de «descendencia común con
modificación»).
Transferencia horizontal de genes
Un medio por el que las bacterias pueden
adquirir
resistencia a los antibióticos es por la transferencia
horizontal de
genes resistentes a los antibióticos. Esta transferencia de
genes de
resistencia es frecuente (Gómez, 1998; Top et al., 2000), y da
cuenta de muchos
casos de resistencia en las bacterias. Sin embargo, la transferencia
horizontal
involucra meramente la transferencia de genes de resistencia que ya
existen en
el mundo de las bacterias.
En tanto que la adquisición horizontal de
genes de
resistencia es «beneficiosa» para las bacterias expuestas a
un antibiótico
determinado, esta transferencia de genes no da cuenta del origen de la
diversa
variedad de estos genes. Como tal, no proporciona un mecanismo
genético para el
origen de ningunos de los genes de resistencia a los
antibióticos existentes en
el mundo biológico. La evolución predice que por medio
del proceso de la
«descendencia común con modificación» puede
explicar el origen y la diversidad
de la vida sobre la tierra; sin embargo, la mera transferencia de genes
preexistentes entre organismos mediante transferencia genética
no proporciona
el necesario mecanismo genético para satisfacer esta
predicción. Tampoco puede
explicar satisfactoriamente el desarrollo simultáneo de ambas
cosas, la
biosíntesis de los antibióticos y los genes de
resistencia —lo cual constituye
un enigma evolutivo (Penrose, 1998). De modo que la transferencia
horizontal de
los genes de resistencia no puede presentarse como un ejemplo apropiado
de
«evolución en la cápsula de Petri».
Mutaciones
Las mutaciones, que se definen como
cualquier
cambio en la
secuencia del ADN (Snyder y Champness, 2003), proporcionan el
único mecanismo
genético conocido para la producción de nuevas
actividades y
funciones genéticas en
el mundo biológico. A la luz de esto, solo las mutaciones tienen
el potencial
de proporcionar un mecanismo para la evolución que explique el
origen de la
resistencia a los antibióticos. Así, solo aquella
resistencia que resulte de
una mutación constituye un ejemplo potencial de
«evolución en acción» (esto es,
de «descendencia común con modificación»).
En presencia de un antibiótico determinado
(o de otros microbicidas),
cualquier mutación que proteja a la bacteria de la cualidad
letal de dicho
compuesto presenta evidentemente un fenotipo «beneficioso».
La selección
natural seleccionará de manera enérgica y bastante
precisa aquellos mutantes
resistentes, lo que se ajusta al marco de una respuesta adaptiva. Pero
el
análisis molecular de dichas mutaciones revela una gran
incongruencia entre la
verdadera naturaleza de la mutación y las demandas de la
teoría de la evolución
(Tabla I).
Tabla I. Fenotipos resultado de
mutaciones conducentes a
resistencias a antibióticos específicos
|
Antibiótico
|
Fenotipo que proporciona la
resistencia
|
|
Actinonina
|
Pérdida
de actividad enzimática
|
|
Ampicilina
|
Respuesta
SOS
que detiene la división celular
|
|
Azitromicina
|
Pérdida
de una proteína reguladora
|
|
Cloranfenicol
|
Reducción
de la formación de una porina o de una proteína reguladora
|
|
Ciprofloxacina
|
Pérdida
de una porina o pérdida de una proteína reguladora
|
|
Eritromicina
|
Reducción
de afinidad a ARNr 23S o pérdida de una proteína
reguladora
|
|
Fluoroquinolonas
|
Pérdida
de afinidad a la girasa
|
|
Imioenema
|
Reducción
de la formación de una porina
|
|
Kanamicina
|
Reducción
de la formación de una proteína de transporte
|
|
Ácido
nalidíxico
|
Pérdida
o desactivación de una proteína reguladora
|
|
Rifampina
|
Pérdida
de afinidad a la ARN-polimerasa
|
|
Estreptomicina
|
Afinidad
reducida al ARNr 16S o reducción de la actividad de transporte
|
|
Tetraciclina
|
Formación
reducida de una porina o de una proteína reguladora
|
|
Zwittermicina
A
|
Pérdida
de fuerza motriz del protón
|
La resistencia bacteriana al antibiótico
rifampina puede
resultar de una mutación común. La rifampina inhibe la
transcripción bacteriana
interfiriendo con la actividad normal de la ARN-polimerasa (Gale et
al.,
1981; Levin y Hatfull, 1993). Las bacterias pueden adquirir resistencia
por una
mutación puntual de la subunidad β de la ARN-polimerasa, que
está
codificada por el gen rpoB
(Enright et al., 1998; Taniguchi et al., 1996; Wang et al., 2001;
Williams et
al., 1998). Esta mutación altera de forma suficiente la
estructura de
la
subunidad β de modo que pierde especificidad para la molécula de
la
rifampina. Como resultado, la ARN-polimerasa deja de tener afinidad
por la rifampina, y
ya no queda afectada por el efecto inhibidor del antibiótico.
De
hecho,
el nivel de resistencia a la rifampina
que puede
adquirir una bacteria de forma espontánea puede ser sumamente
elevado.
En mi
laboratorio obtenemos rutinariamente estirpes mutantes con un nivel de
resistencia varias magnitudes mayor que el de la estirpe silvestre.
Cuando hay rifampina
presente, esta mutación proporciona una clara ventaja para la
supervivencia en
comparación con las células que carecen de estas
mutaciones
específicas. Pero
cada una de estas mutaciones elimina la afinidad de la ARN-polimerasa
por la rifampina. Como tales, estas mutaciones no proporcionan un
mecanismo que expliquen el origen
de la afinidad, sino solo su pérdida.
La resistencia espontánea a las
fluoroquinolonas (como la ciprofloxacina
o la norfloxacina) es también una mutación frecuente en
algunas bacterias. La
diana primaria del antibiótico es el enzima ADN-girasa, que
está formado por
dos proteínas codificadas por los genes gyrA
y gyrB
(Hooper y
Wolfson, 1993). El análisis genético ha descubierto que
la resistencia a esta
clase de antibióticos puede ser resultado de una mutación
puntual en cualquiera
de estos genes (Barnard y Maxwell, 2001; Griggs et al., 1996; Heddle y
Maxwell,
2002; Heisig et al., 1993, Willmott y Maxwell, 1993). Estas mutaciones
de las
subunidades de la girasa parecen ser causa de un cambio de
conformación
suficiente de la girasa de modo que reduce o pierde su afinidad por las
fluoroquinolonas (Figura 1). Una vez más, a pesar de su
naturaleza
«beneficiosa», estas mutaciones no proporcionan un modelo
útil que explique el
origen de la afinidad de la girasa por las fluoroquinolonas.
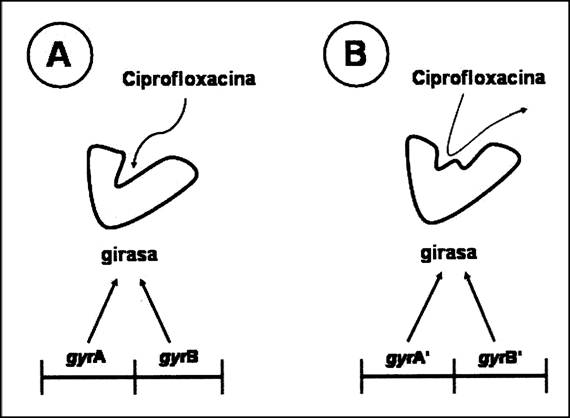
Figura 1. Mecanismo de la
resistencia a la ciprofloxacina.
(A) La
ciprofloxacina interactúa con la girasa, e inhibe su actividad
enzimática. (B)
Una mutación en cualquiera de ambos genes, gyrA o gyrB,
puede
cambiar la estructura que conforma la girasa y reducir la afinidad del
enzima
por la ciprofloxacina. Esto resulta en una incapacidad del
antibiótico para
inhibir la girasa, y la célula se vuelve resistente al
antibiótico.
También la resistencia a la
estreptomicina
puede proceder de
mutaciones bacterianas espontáneas. En este caso, la
estreptomicina bloquea la
síntesis de proteína de la bacteria aparentemente
uniéndose con el segmento del
ARNr 16S del ribosoma e interfiriendo con la actividad del ribosoma
(Carter et
al., 2000; Leclerc et al., 1991). La resistencia al antibiótico
puede surgir
por mutaciones en el gen ARNr 16S, que reduce la afinidad de la
estreptomicina para la molécula 16S (Springer et al., 2001). La
reducción de
unas actividades de transporte específicas de
oligopéptidos lleva también a una
resistencia espontánea frente a diversos antibióticos,
incluyendo la
estreptomicina (Kashiwagi et al., 1998). En estos ejemplos, la
resistencia
surgió como resultado de la pérdida de un componente o
actividad funcionales.
La pérdida de actividad enzimática
puede dar como
resultado la
resistencia al metronidazol. El
metronidazol intracelular se tiene que activar mediante enzimas
antes que pueda servir como agente
antimicrobiano. Esta
activación se consigue mediante el enzima nitrorreductasa NADPH
(Figura
2). Si
el metronidazol no es activado no ejerce un efecto inhibidor sobre la
bacteria. Por ello, si no hay actividad de nitrorreductasa NADPH en la
célula, el
metronidazol permanece inactivo. Puede haber pérdida de la
actividad de
la
reductasa por mutaciones terminadoras o de deleción en rdxA
(Debets-Ossenkopp et al.,
1999; Goodwin et al., 1998;
Tankovic et al., 2000).
Además, la actividad de la nitrorreductasa NADPH se puede
reducir a
causa de una sola mutación de aminoácido (un solo cambio
de aminoácido),
que reduce su
capacidad para activar el metronidazol (Paul et al., 2001). Todas estas
mutaciones resultan en la pérdida de la actividad
enzimática necesaria
para que
el fármaco sea efectivo en la célula, y por ello la
célula se vuelve
resistente
al metronidazol. Pero la pérdida de actividad enzimática
no da ningún
ejemplo
genético de cómo «evolucionó»
originalmente dicho enzima. Por ello, las
mutaciones que proporcionan resistencia frente al metronidazol no
pueden
presentarse como verdaderos ejemplos de «evolución en una
cápsula de Petri».
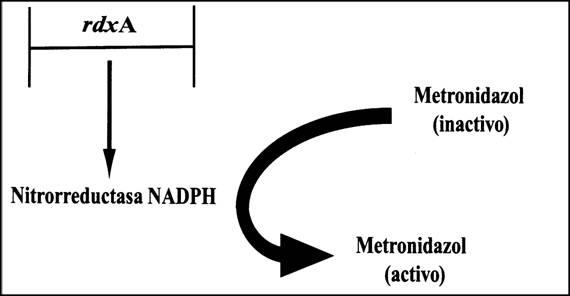
Figura 2. La activación
del agente antimicrobiano,
el
metronidazol.
Después de ser transportado al interior de la célula, el
metronidazol necesita
una modificación estructural para adquirir su forma activa,
antimicrobiana.
Esta activación se logra por la acción del enzima
nitrorreductasa NADPH, que es
producto del gen rdxA. Las
mutaciones del rdxA pueden
impedir la
síntesis de
una nitrorreductasa NADPH con actividad funcional, lo que impide la
activación
del metronidazol.
Una diversidad de bacterias, incluyendo la Escherichia
coli, construyen una bomba de eflujo de resistencia múltiple
a los
antibióticos (MAR) que proporciona a la bacteria una resistencia
a múltiples
tipos de antibióticos, incluyendo la eritromicina, la
tetraciclina, la
ampicilina y el ácido nalidíxico. Esta bomba expulsa el
antibiótico del
citoplasma de la célula, lo que ayuda a mantener los niveles
intracelulares por
debajo de una concentración letal (Grkovic et al., 2002; Okusu
et al., 1996)
(Figura 3). La bomba para MAR está compuesta de las
proteínas MarA y MarB, la
síntesis de las cuales resulta inhibida por la proteína
reguladora, MarR
(Alekshun y Levy, 1999; Poole, 2000) (Figura 3). Las mutaciones que
reducen o
eliminan el control de la represión de MarR resultan en una
sobreproducción de
la bomba de eflujo MarAB, lo que posibilita a la célula expulsar
mayores
concentraciones de antibióticos o de otros agentes bactericidas
(Oethinger et
al., 1998; Poole, 2000; Zarantonelli et al., 1999).
La proteína MarA actúa
también como un regulador
positivo
estimulando una mayor producción de las proteínas MarA y
MarB (Alekshun
y Levy,
1999) [Figura 3]. Además, la proteína MarA inhibe
indirectamente la
producción de la porina, OmpF, un canal en la membrana que
permite la
entrada de algunos
antibióticos en la célula (Cohen et al., 1988). Por ello,
la expresión
aumentada de MarA aumenta la expulsión de antibióticos de
la célula, y
reduce
el transporte de algunos antibióticos al interior de la
célula (Figura
3). Las
mutaciones de marR
que
reducen la expresión o la actividad de la proteína MarR
posibilitarán
así una expresión excesiva de la bomba de eflujo MarAB
(Linde et
al., 2000; Okusu et al., 1996), y proporcionarán una mayor
resistencia
de la
bacteria a diversos antibióticos (Eaves et al., 2004; Hans-Jorg
et al.,
2000;
Notka et al., 2002) [Figura 3]. Los mutantes defectuosos de MarR
presentan
también una mayor tolerancia bacteriana a algunos agentes
químicos
orgánicos,
como el ciclohexano (Aono et al., 1998).
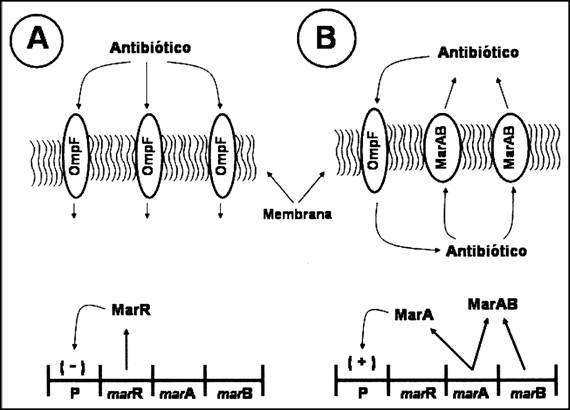
Figura 3.
Bomba de eflujo para resistencia a múltiples fármacos.
(A)
Bacteria sensible a antibióticos. Los antibióticos entran
en la célula a través
de diversos portales, incluyendo la porina OmpF.
La expresión del gen marP
produce la proteína reguladora, MarR. Esta proteína se
une al promotor
(rotulado como P) del operón de resistencia múltiple a
los fármacos, inhibiendo
la expresión de los genes marA y marB.
(B) Bacteria resistente a los
antibióticos. Una mutación de marR
que que reduce la actividad de MarR hace posible que el promotor
funcione
constitutivamente. Ahora se expresan marA
y marB. Estas dos proteínas forman
una bomba de eflujo, que transporta las moléculas de
antibiótico fuera del
citoplasma de la
célula. MarM también se une al promotor
(rotulado como P) y aumenta
la velocidad de transcripción del operón, lo que aumenta
la producción tanto de
MarA como de MarB. Además, la producción de MarA reduce
de forma indirecta la
síntesis de la
porina OmpF,
con lo que se reduce la cantidad de estas porinas en la membrana, La
combinación de un número inferior de porinas para el
transporte de un
antibiótico al interior de la célula, y el aumento de la
cantidad de bombas de
eflujo que eliminan el antibiótico de la célula,
proporciona a la bacteria una
mayor tolerancia a diversos antibióticos.
Las mutaciones que aumentan la producción
de esta bomba de
eflujo hacen posible que estas bacterias sobrevivan la
exposición a diversos
antibióticos. Como tal, esta es una mutación beneficiosa
cuando el antibiótico
está presente en el medio. Sin embargo, una mutación que
es causa de una
pérdida de control de regulación (en este caso de la
proteína represora, MarR)
no ofrece un mecanismo genético que pueda dar cuenta del origen
de este control
regulador.
En otros ejemplos, la resistencia a la
eritromicina puede
también originarse debido a la pérdida de un segmento de
once pares de bases
del gen ARNr 23S (Douthwaite et al., 1985), o por una mutación
que altera la
conformación del ARNr 23S—lo que reduce la afinidad del ribosoma
hacia el
antibiótico (Gregory y Dahlberg, 1999; Vannuffel et al., 1992).
La resistencia
al cloranfenicol se obtuvo por deleción de una región de
12 pares de bases en
el dominio II del gen de la peptidiltransferasa (Douthwaite, 1992). La
resistencia a las cefalosporinas se ha vinculado con una gran
alteración de la
cinética del transporte en membranas que es semejante a las
estirpes
deficientes en porinas (Chevalier et al., 1999). La
resistencia a la actinonina en el Staphylococcus aureus resulta de mutaciones que
eliminan la
expresión del gen fmt (Margolis et al., 2000). La
resistencia a la zwittermicina A en la E. coli está
asociada con la pérdida de fuerza motriz protónica (Stabb
y Handelsoman, 1998).
En el caso del Streptococcus
gordonii, la tolerancia a la penicilina puede involucrar la
pérdida
del control regulador del operón arc (Caldelari et
al., 2000). Y la E.
coli puede sobrevivir a la presencia de las β-lactamas,
como la
ampicilina, deteniendo la división celular, lo que hace a la
célula menos
sensible al efecto letal del antibiótico (Miller et al., 2004).
Estas mutaciones resistentes que se describen
aquí llevan a
la pérdida de un sistema biológico preexistente,
incluyendo la división celular
y la fuerza motriz protónica. Aunque la supervivencia frente al
antibiótico sea un
fenotipo «beneficioso», estas mutaciones no pueden ser un
ejemplo genético de
cómo se originó cada uno de estos sistemas. Como tales,
no proporcionan ningún
medio genético para cumplir las predicciones de
«descendencia con modificación».
La resistencia a otros antibióticos, como
la kanamicina,
puede resultar de la pérdida o reducción de
síntesis de una proteína
transportadora (OppA) [Kashiwagi et al., 1998]. La resistencia a la
ciprofloxacina y a la imipenema puede resultar, al menos en parte, de
una
disminución en la formación de la porina de la membrana
exterior, OmpF (Armand-Lefèvre
et al., 2003;
Hooper
et al., 1987; Yigit et al., 2002). Un aumento en la resistencia al
meropenem y
a la cefepima va también asociado a la pérdida de OmpF y
de otra porina, OmpC
(Yigit et al., 2002). Y el Enterobacter
aerogenes
puede llegar a
hacerse resistente a diversos antibióticos cuando una
mutación reduce en gran
proporción la conductancia de una porina de membrana (Dé
et al., 2001).
Cada una de las resistencias que se describen en
el párrafo
anterior resulta de la reducción o de la pérdida de un
sistema de transporte. Sin
embargo, los mecanismos genéticos necesarios para la
evolución tendrían que dar
cuenta del origen de estos diversos sistemas de transporte. Así,
estas
mutaciones originadoras de la resistencia a los antibióticos no
proporcionan
los cambios genéticos precisos para la «descendencia
común». Al contrario, son
genéticamente incongruentes con las necesidades de la
evolución, siendo que
cada una de ellas involucra la pérdida de una actividad de
transporte
preexistente.
Como grupo, las mutaciones asociadas con la
resistencia a
los antibióticos involucran la pérdida o reducción
de una función o actividad
celular preexistente, esto es, la molécula diana ha perdido una
afinidad hacia
el antibiótico, el sistema de transporte de antibióticos
ha quedado reducido o
eliminado, ha habido reducción o eliminación de un
sistema regulador o de una
actividad enzimática, etc. (Tabla I). Estas no son mutaciones
que puedan dar
cuenta del origen de dichos sistemas y actividades celulares. Aunque
estas
mutaciones pueden ciertamente considerarse como
«beneficiosas» para la
supervivencia de la bacteria cuando está presente un
antibiótico en el medio
ambiente, este beneficio tiene lugar a expensas de una función
previamente
existente. Esto es análogo a eliminar una pared interior de una
casa para
conseguir un comedor más grande. Aunque este comedor mayor pueda
ser deseable
(esto es, beneficioso), el mecanismo de derribo de esta pared no puede
ofrecerse de manera legítima como un ejemplo de cómo se
construyó originalmente
esta pared interior. Igualmente, el beneficio de la supervivencia de
una
mutación es solo una parte de los rasgos genéticos
necesarios para que las
mutaciones puedan dar la «evolución en una cápsula
de Petri». Estas mutaciones
también pueden proporcionar la base genética para una
«descendencia común con
modificación». Aunque esto contradice de forma directa las
pretensiones hechas
por muchos proponentes de la evolución, los datos moleculares
acerca de la
resistencia a los antibióticos son muy claros.
Estas mutaciones tampoco pueden proporcionar un
mecanismo que
siga «evolucionando» el nivel de especificidad o de
actividad de las
proteínas
que se necesitan para la normal función celular. Aunque estas
mutaciones
constituyen unos excelentes ejemplos de adaptación bacteriana,
son en
realidad
lo directamente contrario de los cambios por mutación necesarios
para la
evolución. Sin embargo, estos son precisamente los ejemplos que
los
evolucionistas presentan
como demostraciones verificables del «cambio evolutivo».
Cosa irónica,
estas
mutaciones son en realidad ejemplos verificables de un modelo
creacionista—una
complejidad inicial que pasa por mutación a un nivel de mayor
simplicidad.
La adquisición espontánea de
resistencia a los antibióticos
es designada con frecuencia como una «ganancia» de
resistencia, pero es más apropiado
identificarlo como una pérdida de sensibilidad. Así, la
resistencia a los
antibióticos es resultado de la pérdida de sistemas
previamente existentes en
la célula bacteriana. Está claro que estos cambios no
proporcionan ningún
mecanismo genético para el origen de características
scelulares como la
especificidad
enzimática, la actividad de transporte, la actividad reguladora,
o la afinidad
de las proteínas. Sin embargo, los evolucionistas afirman
insistentemente que las
mutaciones proporcionan un mecanismo genético para el origen de
la actividad
biológica y de una «descendencia común con
modificación», y presentan
repetidamente los tipos de mutación que se acaban de describir
como ejemplos de evolución
en acción.
Costes
en la vitalidad debido a la resistencia a los antibióticos
Aunque las mutaciones que proporcionan
resistencia
a un
antibiótico se pueden considerar «beneficiosas», a
menudo comportan un coste
fisiológico (Andersson y Levin, 1999; Maisnier-Patin et al.,
2002). De hecho, Björkman
et al. (2000) llegan a la conclusión de que la mayoría de
los tipos de
resistencia a los antibióticos impartirán algún
coste biológico al organismo. Por
ejemplo, la resistencia a la rifampina del Mycobacterium
tuberculosis (Billington et al., 1999), de la E. coli (Reynolds,
2000), y del Staphylococcus
aureus (Wichelhaus
et al., 2002) se debían a mutaciones de la ARN-polimerasa que
también redujeron
la capacidad relativa de la mayoría de las estirpes mutantes.
Aunque el coste
biológico comunicado por estos investigadores no era por lo
general muy grave,
era discernible.
Las mutaciones resultantes en una resistencia a la
claritromicina
en el Helicobacter
pylori reducen
la capacidad relativa del organismo (Björkholm et al., 2001). La
resistencia a elevados niveles de
fluoroquinolona por parte de la Salmonella
enterica involucra mutaciones que imparten un elevado coste
biológico al organismo (Giraud et al., 2003). Y las mutaciones
de fusA
que proporcionan
resistencia al ácido fusídico al Staphylococcus sp.
imponen una significativa pérdida de
«capacidad relativa» (Gustafsson
et al., 2003; MacVanin et al., 2000).
La resistencia a la actinonina por parte del S.
aureus va también acompañada de una grave
pérdida de «capacidad» que
da como resultado una disminución considerable en el crecimiento
(Margolis et
al., 2000).
La
resistencia de la E.
coli a
la estreptomicina puede reducir enormemente la velocidad de
biosíntesis de las
proteínas (Zengel et al., 1977).
Y algunas bacterias suspenden la división celular para minimizar
su
sensibilidad a la ampicilina (Miller et al., 2004), lo que
evidentemente reduce
la capacidad global del organismo.
Este coste de la «capacidad relativa»
parece variar
considerablemente, dependiendo tanto del organismo como del
antibiótico. Pero
muchos de los mutantes resistentes que han sido objeto de estudio,
incluyendo
algunos de los que se han mencionado anteriormente, pueden
posteriormente
eliminar algo o mucho del coste sobre la capacidad biológica
mediante retromutaciones
o mutaciones supresoras, que también estabilizan la
mutación (Andersson y
Levin, 1999; Lenski, 1998; Massey et al., 2001). El grado en que una
retromutación restaura la capacidad biológica depende
probablemente del
emplazamiento de la mutación y de si una sola mutación
puede restaurar algo o
todo de la «capacidad» del tipo silvestre.
Es evidente que la capacidad de algunas estirpes
mutantes
queda reducida de forma permanente (algunas veces de forma grave), y
los
evolucionistas, por lo general, han pasado por alto estos efectos en su
precipitación por promover la resistencia a los
antibióticos como «evolución en la cápsula
de Petri». De hecho, a menudo someten a ensayo la capacidad
relativa
de estos mutantes bajo unos parámetros de cultivo muy rigurosos,
que minimizan
la pérdida detectable de capacidad para una mutación
determinada. Por otra
parte, la pérdida de capacidad de algunos mutantes es
despreciable
(especialmente después de retromutaciones). De modo que el
efecto de la
resistencia espontánea sobre la capacidad biológica
bacteriana parece variar de
mutante en mutante. Sin embargo, las mutaciones resistentes imponen
desde luego
un coste biológico por la pérdida de sistemas y
actividades celulares
preexistentes. Este coste biológico no queda compensado por las
retromutaciones
o por las mutaciones supresoras. Aunque dichas mutaciones no siempre
presenten
niveles detectables de reducción de «capacidad», se
levantan como la antítesis
de la «descendencia común con modificación».
Sumario
Con frecuencia se afirma que la resistencia
a los
antibióticos
y a otros microbicidas es una clara demostración de
«evolución en una cápsula de Petri». Sin
embargo, el análisis de los acontecimientos
genéticos que causan
esta resistencia revela que no son congruentes con los acontecimientos
genéticos necesarios para la evolución (definida como
«descendencia común con
modificación»). En lugar de esto, la resistencia que
resulta de la
transferencia horizontal de genes proporciona meramente un mecanismo
para la
transferencia de genes de resistencia previamente existentes. La
transferencia
horizontal no proporciona un mecanismo para el origen de estos genes.
La
mutación espontánea sí que ofrece un potencial
mecanismo genético para el
origen de estos genes, pero este origen nunca se ha podido demostrar.
Al
contrario, todos los ejemplos conocidos de adquisición de
resistencia a los
antibióticos debida a mutación son incongruentes con los
requisitos genéticos
para la evolución. Estas mutaciones dan como resultado la
pérdida de sistemas o
actividades celulares preexistentes, como porinas y otros sistemas de
transporte,
sistemas de regulación, actividades enzimáticas y uniones
de proteínas. La
resistencia a los antibióticos puede también ocasionar
alguna disminución de la
«capacidad relativa» (grave en algunos casos), aunque en el
caso de muchos
mutantes esto quede compensado por una reversión. Sin embargo,
el verdadero
coste biológico es la pérdida de sistemas y actividades
preexistentes. Estas
pérdidas nunca quedan compensadas, a no ser que se pierda la
resistencia, y no
se pueden presentar de forma legítima como ejemplos de un cambio
evolutivo
verdadero.
Referencias
Andersson, D.I., y
B.R. Levin. 1999.
The
biological cost of antibiotic resistance. Current Opinion in
Microbiology 2:489–493.
Aono, R., N.
Tsukagoshi, y M. Yamamoto. 1998.
Involvement of outer membrane protein TolC, a possible member of the mar-sox regulon, in maintenance and improvement of organic
solvent tolerance
of Escherichia
coli K-12. Journal of Bacteriology 180:938–944.
Alekshun, M.N., y
S.B. Levy. 1999. The mar regulon: multiple resistance to
antibiotics and other toxic
chemicals. Trends
in Microbiology 7:410–412.
Armand-Lefèvre,
L.,
V.
Le.on-Guibout, J.
Bredin, F. Barguellil, A. Amor, J.M. Pagès, y M.-H.
Nicolas-Chanoine. 2003.
Imipenem resistance in Salmonella
enterica serovar wien related to porin
loss and
CMY-4 ß-lactamase production. Antimicrobial Agents and
Chemotherapy 47:1165–1168.
Barlow,
M., y B.G. Hall.
2002.
Phylogentic
analysis shows that the OXA â-lactamase genes have been on plasmids for millions of
years. Journal of Molecular Evolution
55:314–321.
Barnard,
F.M., y A. Maxwell.
2001.
Interaction
between DNA gyrase and quinolones: effects of alanine mutations at GryA
subunit
residues Ser83 and Asp87. Antimicrobial
Agents and Chemotherapy 45:1994–2000.
Billington,
O.J., T.D.
McHugh, y
S.H.
Gillespie. 1999. Physiological cost of rifampin induced in vitro in Mycobacterium
tuberculosis. Antimicrobial
Agents and Chemotherapy 43:1866–1869.
Björkholm,
B., I.
Nagaev,
O.G. Berg, D.
Hughes, y D.I. Andersson. 2000. Effects of environment on compensatory
mutations to ameliorate costs of antibiotic resistance. Science 287:1479–1482.
Björkholm,
B., M.
Sjölund, P G. Falk, O.G.
Berg, L. Engstrand, y D.I. Andersson. 2001. Mutation frequency and
biological
cost of antibiotic resistance in Helicobacter pylori. Proceedings of the National
Academy of Science 98:14607–14612.
Caldelari,
I., B. Loeliger,
H.
Langen, M .P.
Glauser, y P. Moreillon. 2000. Deregulation of the arginine deiminase (arc) operon in penicillin-tolerant mutants of Streptococcus
gordonii. Antimicrobial
Agents and Chemotherapy 44:2802–2810.
Carter,
A.P., W. M.
Clemmons, D.E.
Brodersen,
R.J. Morgan-Warren, B.T. Wimberly, y V. Ramakrishnan. 2000. Functional
insights
from the structure of the 30S ribosomal subunit and its interaction
with antibiotics. Nature 407:340–348.
Chevalier,
J., J.-M.
Pagès, y
M. Malléa. 1999. In vivo modification of porin activity conferring antibiotic
resistance to Enterobacter aerogenes. Biochemical
and Biophysical Research Communications 266:248–251.
Cohen,
S.P., L. M. McMurry,
y S.B.
Levy. 1988. marA locus causes decreased expression of OmpF porin in
multiple-antibiotic-resistant (Mar) mutants of Escherichia coli. Journal
of Bacteriology 170:5416–5422.
Darwin,
C. 1936. Origin of Species
and The Descent of Man (Modern Library
Reprint Edition) Random House, New York.
De,
E., A. Baslé, M.
Jaquinod,
N. Saint, M.
Malléa, G. Molle, y J.-M Pagès. 2001.
A new mechanism of antibiotic
resistance in Enterobacteriaceae induced by a structural modification
of the
major porin. Molecular
Microbiology 41:189–198.
Debets-Ossenkopp,
Y.J.,
R..G.J.
Pot, D.J. van
Westerloo, A. Goddwin, C.M.J.E. Vandenbroucke-Grauls, D.E. Berg, P.S.
Hoffman, y
J.G. Kusters. 1999. Insertion of mini-IS605 and deletion of adjacent
sequences
in the nitroreductase (rdxA) gen causes metronidazole resistance in Helicobacter
pylori NCTC11637. Antimicrobial
Agents and Chemotherapy 43:2657–2662.
Dillon,
L.S. 1978. Evolution:
concepts and consequences, C.V. Mosby,
St. Louis, MO.
Douthwaite,
S. 1992.
Functional
interactions
within 23S rRNA involving the peptidyltransferase center. Journal of
Bacteriology 174:1333–1338.
Douthwaite,
S., J.B. Prince, y
H.F.
Noller. 1985. Evidence for functional
interaction between domains II and V
of 23S ribosomal RNA from an erythromycin-resistant mutant. Proceedings of
the National Academy of Science 82:8330–8334.
Enright,
M., P. Zawadski, P.
Pickerill, y C.G.
Dowson. 1998. Molecular evolution of rifampicin resistance in Streptococcus
pneumoniae. Microbial
Drug Resistance 4:65–70.
Eaves, D.J., V.
Ricci, y L.J.V. Piddock. 2004.
Expression of acrB, acrF, acrD, marA, and soxS in Salmonella
enterica serovar typhimurium: role in
multiple
antibiotic resistance. Antimicrobial
Agents and Chemotherapy 48:1145–1150.
Gale,
E.F., E. Cundliffe,
P.E.
Reynolds, M.H.
Richmond, y M.J. Waring. 1981. The Molecular Basis of
Antibiotic Action. John Wiley & Sons,
New York.
Giraud,
E., A. Cloeckaert, S.
Baucheron, C.
Mouline, y E. Chaslus-Dancla. 2003. Fitness
cost of
fluoroquinolone resistance in Salmonella enterica serovar Typhimurium.
Journal
of Medical Microbiology 52:697–703.
Gómez,
L.R. 1998.
Evolution
of bacterial
resistance to antibiotics during the last three decades. International
Microbiology 1:279–284.
Goodwin,
A., D. Kersulyte,
G.
Sisson, S.J.O.V.
van Zanten, D.E. Berg, y P.S. Hoffman. 1998. Metronidzole resistance in
Helicobacter pylori is due to null mutations in a gen (rdxA) that
encodes an
oxygen-insensitive NADPH nitroreductase. Molecular Microbiology
28:383–393.
Gregory,
S.T., y A.E.
Dahlberg.
1999.
Erythromycin resistance mutations in ribosomal proteins L22 and L4
perturb the
higher order structure of 23 S ribosomal RNA. Journal of Molecular
Biology
289:827–834.
Griggs,
D.J., K. Gensberg, y
L.J.
Piddock.
1996. Mutations in gyrA gen of quinolone-resistant Salmonella serotypes
isolated from humans and animals. Antimicrobial Agents and Chemotherapy
40:1009–1013.
Grkovic,
S., M.H. Brown, y
R.A.
Skurray. 2002.
Regulation of bacterial drug export systems. Microbiology and Molecular
Biology
Reviews 66:671–701.
Gustafsson,
I., O. Cars, y
D.I.
Andersson.
2003. Fitness of antibiotic resistant Staphylococcus epidermidis
assessed by
competition on the skin of human volunteers. Journal of Antimicrobials
and
Chemotherapy 52;258–263.
Hans-Jörg,
L., F.
Notka, M.
Metze, B.
Kochanowski, P. Heisig, y N. Lehn. 2000. Antimicrobial Agents and
Chemotherapy
44:1865–1868.
Heddle,
J., y A. Maxwell.
2002.
Quinolone-binding pocket of DNAgyrase: role of GryB. Antimicrobial
Agents and
Chemotherapy 46:1805–1815.
Heisig,
P., H. Schedletzky, y
H.
Falkenstein-Paul. 1993. Mutations in the
gyrA gen of a
highly fluoroquinolone-resistant clinical isolate of Escherichia coli.
Antimicrobial Agents and Chemotherapy 37:696–701.
Hooper,
D.C., y J.S.
Wolfson.
1993. Quinoline
Antimicrobial Agents. ASM Press, Washington, DC.
Hooper,
D.C., J.S. Wolfson,
E. Y.
Ng, y M. N.
Swartz. 1987. Mechanisms of action and resistance to ciprofloxacin.
American
Journal of Medicine 82:4A:12–20.
Johnson,
G.B. 2000. The
Living
World. McGraw-Hill, New York.
Kashiwagi,
K., M.H. Tsuhako,
K.
Sakata, T.
Saisho, A. Igarashi, S.O.P. daCosta, y K. Igarashi. 1998. Relationship
between
spontaneous aminoglycoside resistance in Escherichia coli and a
decrease in
oligopeptide binding protein. Journal of Bacteriology 180:5484–5488.
Leclerc, D., P.
Melancon, y L. Brakier-Gingras. 1991. Mutations in
the 915 region of
Escherichia coli 16S
ribosomal
RNA reduce the binding of streptomycin to the ribosome. Nucleic Acid
Research
19:3973–3977.
Linde,
H.-J., F. Notka, M.
Metz, B.
Kochanowski,
P. Heisig, y N. Lehn. 2000. In vivo
increased resistance to Ciprofloxacin in Escherichia coli associated with deletion of the C-terminal part of MarR. Antimicrobial
Agents and
Chemotherapy 44:1865–1868.
Lenski,
R.E. 1998. Bacterial
evolution and the
cost of antibiotic resistance. International Microbiology 1:265–270.
Levin,
M.E., y G. F. Hatfull. 1993. Mycobacterium
smegmatis RNA polymerase: DNA
supercoiling, action of rifampicin and mechanism
of rifampicin resistance. Molecular
Microbiology 8:277–285.
MacVanin,
M., U. Johanson,
M.
Ehrenberg, y D.
Hughes. 2000. Fusidic acid-resistant EF-G perturbs the accumulation of
ppGpp. Molecular Microbiology 37:98–107.
Maisnier-Patin,
S., O.G.
Berg, L.
Liljas, y
D.I. Andersson. 2002. Compensatory adaptation to the deleterious
effects of
antibiotic resistance in Salmonella
typhimurium. Molecular Microbiology 46:355–366.
Margolis,
P.S., C.J.
Hackbarth,
D.C. Young, W.
Wang, D. Chen, Z. Yuan, R. White, y J. Trias. 2000. Peptide deformylase
in Staphylococcus
aureus: resistance to inhibition is
mediated by mutations in the
formyltransferase gen. Antimicrobial
Agents and Chemotherapy 44:1825–1831.
Massey,
R.C., A. Buckling, y
S.J.
Peacock.
2001. Phenotypic switching of antibiotic resistance circumvents
permanent costs
in Staphylococcus
aureus. Current Biology 11:1810–1814.
Miller,
C., L.E. Thomsen, C.
Gaggero, R.
Mosseri, H. Ingmer, S.N. Cohen. 2004. SOS response induction by
ß-lactams and
bacterial defense against antibiotic lethality. Science 305:1629–1631.
Miller,
K.R. 1999. Finding Darwin’s
God. Harper Collins, New York.
Mortlock,
R.P. 1984. Microorganisms as
Model
Systems for Study-ing Evolution. Plenum
Press, New York.
Notka,
F., H.-J. Linde, A.
Dankesreiter, H.-H.
Niller, y N. Lehn. 2002. A
C-terminal
18 amino acid deletion in MarR in a clinical isolate of Escherichia coli reduces MarR binding properties and increases the MIC of
ciprofloxacin. Journal
of Antimicrobial Chemotherapy 49:41–47.
Oethinger,
M., I. Podglajen,
W.V.
Kern, y S.T.
Levy. 1998. Overexpression of the marA and soxS regulatory gen in clinical topoisomerase mutants of Escherichia coli. Antimicrobial
Agents and Chemotherapy 42:2089–2094.
Okusu, H., D. Ma, y
H. Nikaido. 1996.
AcrAB
efflux pump plays a major role in the antibiotic resistance phenotype
of Escherichia
coli multiple-resistance (Mar) mutants. Journal of
Bacteriology 178:306–308.
Patterson,
C. 1978. Evolution. Cornell University Press,
Ithaca, NY.
Paul,
R., S. Postius, K.
Melchers, y
K.P.
Schäfer. 2001. Mutations of the Helicobacter
pylori genes rdxA and pbp1 cause resistance against metronidazole and amoxicillin.
Antimicrobial
Agents and
Chemotherapy 45:962–965.
Penrose,
E. 1998. Bacterial
resistance to
antibiotics—a case of un-natural selection. Creation Research
Society Quarterly 35:76–83.
Poole,
K. 2000.
Efllux-mediated resistance to fluoroquinolones in gram-negative
bacteria. Antimicrobial Agents and
Chemo-therapy 44:2233–2241.
Reynolds,
M.G. 2000.
Compensatory
evolution in
rifampin-resistant Escherichia
coli. Genetics 156:1471–1481.
Springer,
B., Y.G. Kidan, T.
Prammananan, K.
Ellrott, E. C. Böttger, y P. Sander. 2001. Mechanisms of
streptomycin
resistance: selection of mutations in the 16S rRNA gen conferring
resistance. Antimicrobial Agents and
Chemotherapy 45:2877–2884.
Stabb,
E.V., y J. Handelsman. 1998. Genetic
analysis of zwittermicin A resistance in Escherichia coli: effects on membrane potential and RNA polymerase. Molecular
Microbiology 27:311–322.
Snyder,
L., y W. Champness.
2003. Molecular
Genetics of Bacteria. ASM Press,
Washington, DC.
Taniguchi,
H. H. Aramaki, Y.
Nikaido, Y.
Mizuguchi, M. Naka-mura, T. Koga, y S. Yoshida. 1996. Rifampicin
resistance and
mutation of the rpoB gen in Mycobacterium
tuberculosis. FEMS
Microbiological Letters 144:103–108.
Tankovic,
J., D. Lamarque,
J.-C.
Delchier, C.-J.
Soussy, A. Labigne, y P.J. Jenks. 2000.
Frequent
association between alteration of the rdxA gen and
metronidazole resistance in French and North African isolates Helicobacter
pylori. Anti-microbial
Agents and Chemotherapy 44:608–613.
Top,
E.M., Y.
Moënne-Loccoz,
T. Pembroke, y
C.M. Thomas. 2000. Phenotypic traits conferred by plasmids. In Thomas,
C.M.
(editor), The
Horizontal Gen Pool, pp. 249–285. Harwood
Academic, Amsterdam, The Netherlands.
Vannuffel, P., M. Di
Giambattista, E.A. Morgan,
y C.
Cocito. 1992. Identification of a single base
change in ribosomal RNA
leading to erythromycin resistance. Journal of Biological
Chemistry 267:8377–8382.
Wang,
G., T. J. M. Wilson,
Q.
Jiang, y D.E.
Taylor. 2001. Spontaneous mutations that confer antibiotic resistance
in Helicobacter
pylori. Antimicrobial
Agents and Chemotherapy 45:727–733.
Wichelhaus,
T.A., B.
Böddinghaus, S. Besier, V.
Schäfer, V. Brade, y A. Ludwig. 2002.
Biological cost
of rifampin from the perspective of Staphylococcus aureus.
Antimicrobial
Agents and Chemotherapy 46:3381–3385.
Williams,
D.L., L. Spring,
L.
Collins, L.P.
Miller, L.B. Heifets, P.R.J. Gangadharam, y T.P. Gillis. 1998.
Contribution of rhoB mutations to development of reifamycin cross-resistance
in Mycobacterium
tuberculosis. Antimicrobial
Agents and Chemotherapy 42:1853–1857.
Willmott,
C.J.R., y A.
Maxwell.
1993. A single point mutation in the DNA gyrase A protein greatly
reduces the binding of
fluoroquinolones to the gyrase-DNA complex. Antimicrobial
Agents and Chemotherapy 37:126–127.
Yigit,
H., G. J. Anderson,
J. W.
Biddle, C. D.
Steward, J. K. Rasheed, L.L. Valera, J.E. McGowan, y F.C. Tenover.
2002.
Carbapenem resistance in a clinical isolate of Enterobacter
aerogenes is associated with decreased
expression of OmpF and OmpC porin
analogs. Antimicrobial
Agents and Chemotherapy 46:3187–3822.
Zarantonelli, L., G.
Borthagaray, E.-H. Lee, y
W. M.
Shafer. 1999. Decreased azithromycin susceptibility
of Neisseria
gonorrhoeae due to mtrR mutations. Antimicrobial
Agents and Chemotherapy 43:2468–2472.
Zengel,
J.M., R. Young, P.P.
Dennis, y M.
Nomura. 1977. Role of ribosomal protein S12 in peptide chain
elongation:
analysis of pleitropic, streptomycin-resistant mutants of Escherichia coli. Journal
of
Bacteriology 129:1320–1329.






